不同环境样本类型对蚌类环境DNA监测的差异研究
时间:2023-04-20 21:00:02 来源:千叶帆 本文已影响人
雷 姚 周春花, 欧阳珊, 吴小平,
(1. 南昌大学生命科学学院, 南昌 330031; 2. 鄱阳湖环境与资源利用教育部重点实验室, 南昌 330031; 3. 江西省流域生态演变与生物多样性重点实验室, 南昌 330031)
淡水生态系统为人类的文明发展提供了宝贵资源, 然而却存在生物多样性丧失和调查研究不足的现状[1]。物种是生物多样性的重要组成成分, 进行种质资源调查和监测是保护生物多样性的重要基石[2], 在全球生物多样性丧失日益严重的背景下,对生物多样性现状全面了解的需求尤为紧迫[3]。长江是我国淡水资源的宝库, 但多年来受人类活动影响, 长江生物多样性持续下降, 水生态保护形势严峻[4]。农业农村部于2020年1月1日颁布的《长江流域重点水域禁捕和建立补偿制度实施方案》提出长江要开始为期10年的全面禁渔, 且越来越多的大江大河开始加大禁渔力度、扩大禁渔区的范围。在禁渔条件下, 许多传统的监测水生生物的方法难以适应新的监测要求, 因此创新水生生物资源监测方法是重要的科学问题。
蚌类是大型底栖动物, 是生态系统中最重要的生物类群之一。一方面, 它们被如底层鱼类的小型动物和一些鸟类所食用; 另一方面, 它们通过滤食水体中的营养物质、有机物和浮游生物从而达到净化水体的效果。蚌类在食物网中扮演着重要的角色, 栖息于湖泊或河流的淡水双壳类消失将会导致一系列的生态问题[1]。最近几十年来, 全世界范围内蚌类种群密度和种类丰富程度等呈现明显的下降趋势, 部分种群自我恢复的能力远低于其物种数量减少的速度, 导致一些物种濒临灭绝或已灭绝[2]。蚌类作为最易受威胁的淡水生物类群之一, 自20世纪90年代以来, 其种群呈明显的衰退趋势[3]。蚌类的分布具有明显的地域特征, 北美蚌类特有种多集中于美国, 而亚洲蚌类最丰富的国家则是中国, 且集中分布在长江中下游地区[4]。鄱阳湖是我国最大的淡水湖泊, 有着丰富的蚌类资源, 由于人类活动如挖沙、水体污染和过度捕捞等对蚌类的生存造成了威胁, 也影响了湖泊的正常生态功能[5]。鄱阳湖的蚌类资源最早由Heude[6]进行过采集和形态鉴定, 之后林振涛[7]、张玺等[8]和刘月英等[9]陆续对鄱阳湖蚌类进行了区系调查, 吴小平等[10]和欧阳珊等[11]也分析了鄱阳湖蚌类的分布及资源现状。2009年的调查数据显示鄱阳湖有蚌类42种, 其中包括中国特有种32种[12]。2016—2017系统调查鄱阳湖大型底栖动物数据表明鄱阳湖有蚌类24种[13]。通过与历史数据比较, 发现鄱阳湖的蚌类物种丰富度和优势物种均显著减少。
虽然传统的调查方法在调查蚌类多样性方面发挥着不可替代的作用, 但也存在如所需时间长、依赖专家经验、环境破坏性大、稀有物种调查不易、与禁渔政策相违背等诸多问题[14]。环境DNA宏条形码(Environmental DNA metabarcoding,eDNA metabarcoding)技术直接从环境样本(如水、沉积物和土壤等)中提取DNA并使用针对目标物种的通用引物进行扩增, 利用PCR和高通量测序(High-throughput sequencing, HTS)等技术, 可在较短时间内实现对多个目标物种的鉴定[15]。Prié等[16]利用环境DNA宏条形码评估了西古北界淡水双壳类两个科的生物多样性。Klymus等[17]利用环境DNA宏条形码方法检测了美国东南部的蚌科和珍珠蚌科。陈金萍等[18]筛选出的宏条形码引物被证明在蚌类多样性研究中的有效性。
环境DNA调查信息丰富, 可以补充其他生物多样性监测方法的不足, 不同环境样本类型会影响物种可检测性和群落结构[19]。Jennifer等[20]将水体样本与沉积物样本检出的鱼类物种数与传统鱼类调查方法进行了比较, 结果表明与沉积物相比, 水体样本中提取的环境DNA所反映的物种数量较多, 且更能反映鱼类群落的特征。Sakata等[21]通过对不同环境样本类型进行比较发现, 水体样本和沉积物样本检出的鱼类物种数量相当, 但其物种组成之间差异显著。Kusanke等[22]通过检测水体样本和沉积物样本中的濒危物种Misgurnus fossilis, 发现目标生物在沉积物中的环境DNA浓度高于水体, 这表明对于M. fossilis这种一生大部分时间都隐藏在沉积物中的物种而言, 采集沉积物样本更适合。钱瑭毅等[23]通过采集表层水、中层水和底层水来分析在越冬洄游时中国对虾(Fenneropenaeus chinensis)的分布情况, 结果表明, 在自然水体中, 中国对虾的环境DNA呈现出底层浓度高于表层浓度的垂直分布规律。Moyer等[24]为了确定环境DNA检测时的有效水层, 以Hemichromis letourneuxi为目标生物, 结果表明在进行环境DNA检测的最佳水层为表层水, 底层水次之。目前, 环境DNA宏条形码在蚌类的研究中多采集表层水[16,17], 不同的环境样本类型对蚌类多样性及群落结构的差异研究还很少见。蚌类营底栖生活, 因此我们提出假设: 环境DNA宏条形码在蚌类多样性的研究中, 沉积物样本检测的效果会优于水体样本, 底层水的效果会优于表层水。
本研究结合传统蚌类调查方法, 探索使用环境DNA宏条形码技术时, 不同环境样本类型如何影响蚌类物种的可检测性, 并对不同环境样本类型得到的蚌类多样性及群落结构差异进行探究。旨在探讨环境DNA宏条形码技术应用于蚌类多样性及群落结构的研究是否可行, 并为后续蚌类环境DNA监测时的取样策略的选择提供依据。
1.1 样本采集
本研究分别于2021年1月19—21日(冬季)和4月28—30日(春季)在鄱阳湖采集环境水样和沉积物样本并进行蚌类传统方法的采集, 共设置采样点17个,其中环境DNA采样点6个, 17个采样点均进行传统方法的采集(图 1)。在每个环境DNA采样点分别采集表层水(水面下约0.5 m)和底层水(湖底上方约1 m)各1 L于灭菌广口瓶中, 冰上保存并尽快运回实验室过滤, 且表层水和底层水在每个采样点均做3次重复。使用彼得森采泥器(面积为1/16 m2)采集沉积物, 取0.5 g沉积物于50 mL样品瓶中, 低温保存直至样本处理。使用边长为0.6 m的自制蚌耙在17个传统方法采样点, 将蚌耙拉绳的一端固定于船尾, 匀速拖拉50 m, 打捞约30 m2内的蚌类。使用稀释的商业漂白剂溶液(>0.1%次氯酸钠溶液)对采样步骤中使用的所有设备进行灭菌以防污染。使用多参数水质仪(YSI650-MDS)记录各采样点的溶解氧(DO)、水温(WT)、酸碱度(pH)和叶绿素a(Chl.a)等环境参数。使用透明度盘进行透明度(SD)的测定。使用采泥器进行水深(WD)的估算。每个样点采集水样1 L, 交于江西省九江市星子县环境监测站测定总氮(TN)和总磷(TP)。

图1 鄱阳湖采样图Fig. 1 Sampling map of Poyang Lake
1.2 样品处理
水样采集后于当天6h之内用0.45 μm的混合纤维素滤膜(天津津腾/JINTENG, 中国)对水样进行真空泵抽滤, 以达富集环境DNA的目的, 将各个样品的滤膜置于1.5 mL离心管中于-20℃冰箱冷冻保存。抽滤过程中所需设备及器材进行无菌处理, 并使用超纯水作为阴性对照。将沉积物样本从-80℃冰箱拿出置于4℃消解, 将消解好的沉积物称量0.25 g于1.5 mL离心管中, 简短离心使其达到水泥分离的状态。标本鉴定主要依据《中国经济动物志: 淡水软体动物》[9]。在实验室对每个样点的蚌类物种进行计数和称重。测量完的活体标本用95%的乙醇固定保存用于后续的分子实验。
1.3 环境DNA提取与扩增
使用试剂盒DNeasy®Blood & Tissue Kit(Qiagen, Venlo, the Netherlands)并按照说明书进行滤膜DNA提取。使用试剂盒DNeasy®PowerSoil®Pro Kit Handbook(Qiagen, Venlo, the Netherlands)并按照说明书进行沉积物DNA提取。最后使用Qubit(Thermo Fisher Scientific, 中国)测定DNA浓度和质量。
使用蚌类的通用引物16SPp-F: 5′-TGAGCGTG CTAAGGTAGC-3′和16SPp-R: 5′-GCGGGGTC TTTTYGTCT-3′[18], 对环境样本(水体样本和沉积物样本)的16S rRNA区域进行PCR扩增, 每个样本做3个重复扩增, 扩增片段长度约为147 bp。PCR反应采用20 μL体系, 其中包含: 5×FastPfu Buffer 4 μL,2.5 mmol/L dNTPs 2 μL, Forward Primer(5 μmol/L)0.8 μL, Reverse Primer(5 μmol/L)0.8 μL, FastPfu Polymerase 0.4 μL, Template DNA 10 ng, 补ddH2O至20 μL。PCR反应程序: 95℃预变性5min, 29个循环包括: 95℃变性30s, 55℃退火30s, 72℃延伸45s;最后72℃延伸10min, 4℃保存。使用ddH2O为模板设置PCR阴性对照, PCR阴性对照扩增结果未见异常。PCR扩增后所得产物经2%琼脂糖凝胶电泳检测后, 送至上海凌恩生物科技有限公司进行高通量测序。
1.4 高通量测序与质控
选择Illumina Miseq平台进行高通量测序。对下机数据进行筛选和过滤后得到有效序列。使用QIIME软件将有效序列聚类为可操作分类单元(Operational taxonomic unit, OTU), 获得OTU代表序列。OTU代表序列使用本地和公共数据库相结合的方式进行物种注释分析。本地数据库由鄱阳湖历史记录蚌类37种(物种信息见附录1)的线粒体全基因组序列或16S rRNA序列构成, 选择Nt数据库(https://blast.ncbi.nlm.nih.gov/)作为公共数据。物种注释标准依据Valdivia-Carrillo等[25], 以97%—100%确定为种; 94%—97%确定为属; 91%—94%确定为科; 88%—91%确定为目; <88%确定为纲。
1.5 数据分析
数据分析主要有: 物种组成分析、多样性分析和相关性Heatmap分析。
物种组成分析物种组成分析选用相似水平为97%的OTU样本表。其中, 相对序列丰度计算了不同的蚌类物种在各采样点的reads之和。蚌类生物量计算了每个采样点约30 m2断面内活体蚌类的重量之和。
Alpha多样性分析Alpha多样性选取Chao1指数、物种丰富度指数、香农指数和辛普森指数进行分析。其中, Chao1指数和Richness指数用于群落丰富度的估算。Shannon指数和Simpson指数则用于估算群落多样性。Chao1指数由Chao(1984)最早提出, Chao1值越大, 代表群落物种越丰富。
式中,SChao1= 估计的OTU数;Sobs=实际观测到的OTU数;n1=只含有一条序列的OTU数目;n2=只含有两条序列的OTU数目。
物种丰富度指数(Species richness)是指群落中丰度大于0的物种数之和。Species richness指数值越大, 说明群落物种种类越丰富。
式中,S为物种丰富度指数;n为个体数(丰度)大于0的物种类型总数。
香农指数(Shannon index)是用来估算样本中生物多样性的指数之一。Shannon值越大, 说明群落多样性越高。
式中,Sobs= 实际观测到的OTU数;ni=第i个OTU所含的序列数;N=所有的序列数。
辛普森指数(Simpson index)是用于估算样本中生物多样性的指数之一。Simpson指数值越大, 说明群落多样性越低。
式中,Sobs= 实际观测到的OTU数;ni=第i个OTU所含的序列数;N=所有的序列数。
Beta多样性分析Beta多样性主要通过非度量多维尺度分析(Non-metric multidimensional scaling, NMDS)对群落结构进行研究。以胁迫系数(stress)来衡量NMDS结果的优劣, stress值越小越好,当stress<0.05时为拟合极好, stress<0.1时为拟合较好, stress<0.2时为拟合一般, stress>0.3为拟合较差。选用相似性分析(Analysis of Similarities, ANOSIM)来验证组内差异是否显著小于组间(两组或多组)差异, 从而判断分组是否有意义。
相关性Heatmap分析相关性Heatmap分析是以热图的形式呈现出物种与环境因子之间的相关性, 可观察物种与环境因子之间相关性的正负程度。*为P<0.05, **为P<0.01, ***为P<0.001, 不同的颜色代表不同的R值, 热图最右侧的色卡是不同R值的颜色分区。
2.1 鄱阳湖蚌类物种组成分析
本研究基于Illumina PE250测序对36个样本进行高通量测序, 经质控和过滤, 共得到有效序列22320294条, 平均序列长度115 bp, 在97%的相似水平下进行OTU聚类, 共得到33686个OTU。本研究共注释到蚌类33种, 其中30种是鄱阳湖记录种(表 1)。从季节来看, 冬季注释到蚌类28种, 春季注释到蚌类32种。从环境样本来看, 表层水注释到蚌类32种,底层水注释到蚌类32种, 沉积物注释到蚌类28种。传统方法在17个采样点共采集蚌类18种, 其中冬季采集到17种, 春季采集到11种。基于环境DNA宏条形码技术能检测出传统方法采集到的所有蚌类物种, 其中, 表层水除剑状矛蚌未检测到外, 其余传统方法采集到的蚌类物种均可检测到; 底层水能检测到传统方法采集到的所有物种; 沉积物中除剑状矛蚌、卵形尖嵴蚌、舟行无齿蚌和猪耳弓背蚌未检测到外, 其余传统方法采集到的蚌类物种均可检测到。本次测序结果相对序列丰度最高的为橄榄蛏蚌(1754829), 其次为圆顶珠蚌(579313)。传统方法采集到的蚌类生物量最高的为洞穴丽蚌(84.29 g/m2),其次为扭状矛蚌(65.26 g/m2; 表 2)。从环境DNA宏条形码中获得的相对序列丰度与蚌类生物量之间不存在正相关关系。
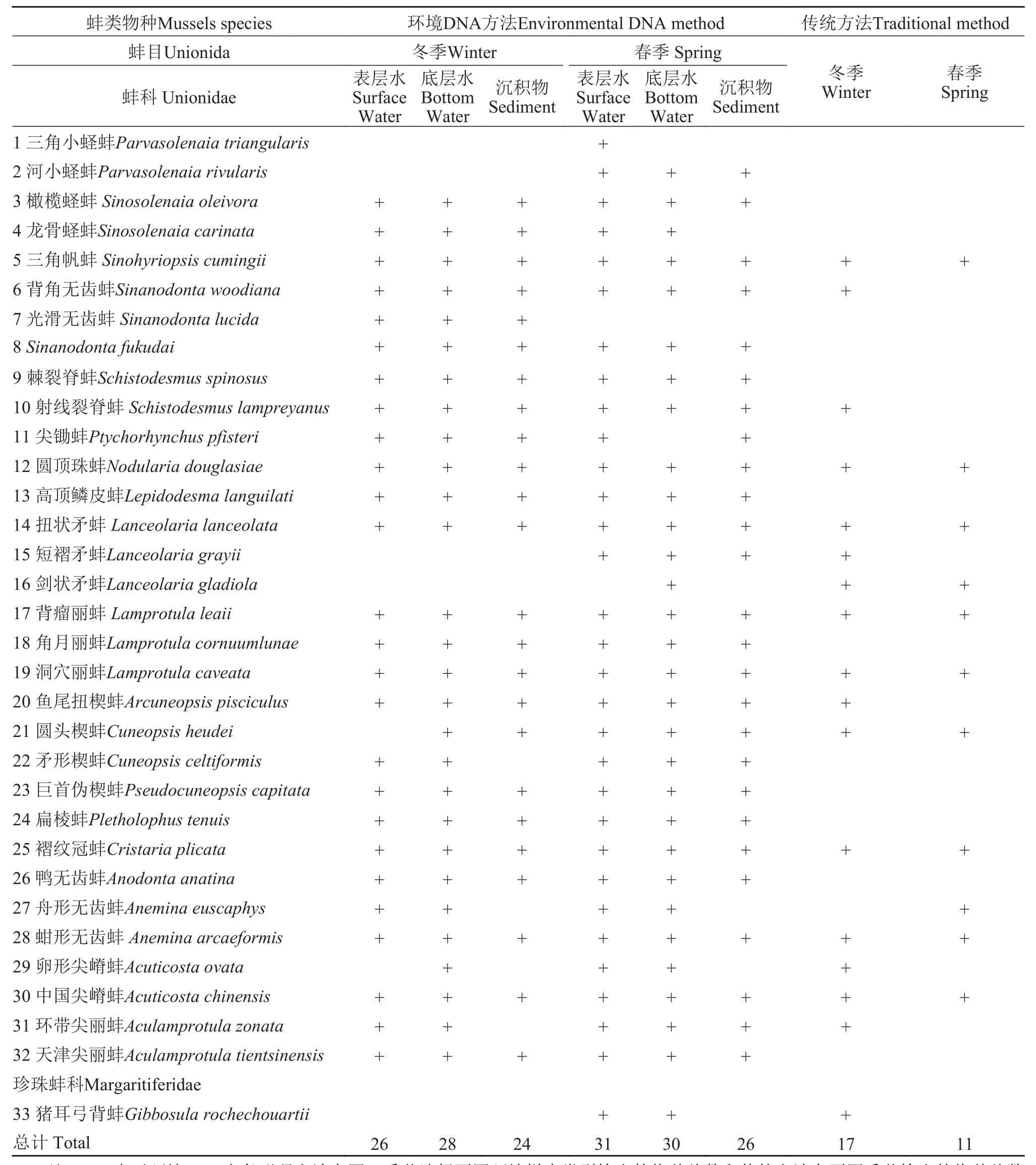
表1 环境DNA方法检出的蚌类与传统方法采集的蚌类Tab. 1 Mussels detected by environmental DNA methods and collected by traditional methods

表2 环境DNA相对序列丰度与传统方法蚌类生物量对比Tab. 2 Relative sequence abundance of environmental DNA species compared with biomass of mussels by traditional methods
蚌类动物线粒体的遗传方法是双单亲遗传, 存在母系和父系来源的序列。本研究使用的引物能分辩母系和父系来源的序列。有的物种只可分辨父系来源的序列如洞穴丽蚌, 有的物种只可分辨母系来源的序列有: 高顶鳞皮蚌、蚶形无齿蚌、中国尖嵴蚌、龙骨蛏蚌、角月丽蚌、褶纹冠蚌、尖锄蚌、天津尖丽蚌、矛形楔蚌、环带尖丽蚌、短褶矛蚌、光滑无齿蚌、卵形尖嵴蚌、舟形无齿蚌和三角小蛏蚌。有的物种可分辨母系和父系来源的序列。在能分辩母系和父系来源的序列物种中, 有的父系reads多, 有的母系reads多(表 3)。

表3 环境DNA方法鉴定的蚌类F型和M型reads数Tab. 3 Number of F-type and M-type reads identified by environmental DNA method
2.2 鄱阳湖蚌类物种Alpha多样性分析
基于环境DNA宏条形码的鄱阳湖蚌类Alpha多样性从季节上看, 虽然春季的Chao1指数和Richness指数均高于冬季, 但春季和冬季Alpha多样性水平无显著性差异(P=0.282; 图 2)。从环境样本类型来看, 表层水的Chao1指数和Richness指数显著高于沉积物的, 底层水的Chao1指数和Richness丰富度指数极显著高于沉积物的。底层水的Shannon多样性指数和Simpson指数和沉积物的均有显著性差异。表层水和底层水多样性水平没有差异(图 3)。
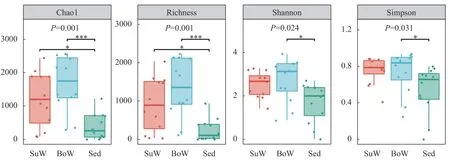
图3 不同环境样本类型Alpha多样性水平差异检验箱线图Fig. 3 Box plot of Alpha diversity level difference test for different environmental sample types
2.3 鄱阳湖蚌类物种Beta多样性分析
基于环境DNA宏条形码的鄱阳湖蚌类结果进行Bray-Curtis NMDS分析表明冬季与春季的蚌类群落相似性的Stress值为0.1509, 进一步地ANOSIM分析显示:R值>0, 即组间差异大于组内差异, 分组有意义,P<0.01, 即春季和冬季的环境DNA宏条形码注释到的鄱阳湖蚌类群落结构差异达到极显著性水平(R=0.252,P=0.001; 图 4)。从环境样本类型来看, Stress值为0.1509, ANOSIM分析中,R值大于0, 整体P值小于0.01, 表明不同环境样本类型的整体群落结构组成具有极显著差异, 进一步的ANOSIM分析表明表层水和底层水的环境DNA注释到的鄱阳湖蚌类群落结构差异尚未达到显著性水平(R=0.002,P=0.398); 表层水和沉积物的蚌类的群落结构有显著性差异(R=0.148,P=0.032); 底层水和沉积物的蚌类的群落结构差异达到极显著性水平(R=0.272,P=0.007; 图 5)。

图4 冬季和春季注释到的蚌类群落NMDS排序Fig. 4 NMDS ordination of mussel communities annotated in winter and spring
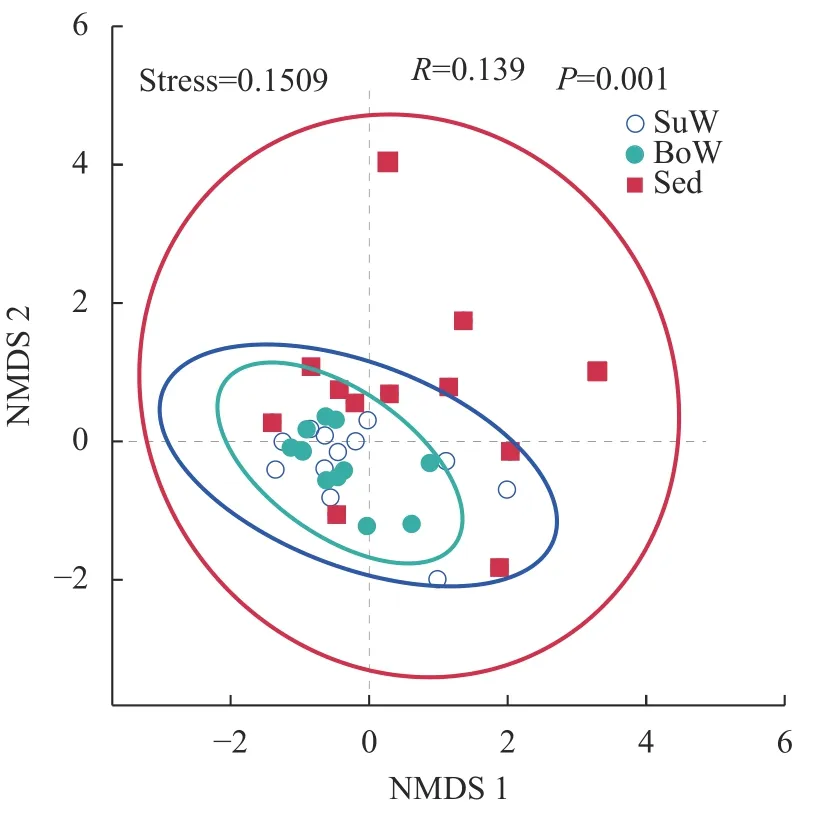
图5 表层水、底层水和沉积物注释到的蚌类群落NMDS排序Fig. 5 NMDS ordination of mussel communities annotated by surface water, bottom water and sediment
2.4 基于环境DNA宏条形码技术的鄱阳湖蚌类群落结构与环境因子的关联分析
基于环境DNA宏条形码技术检测到的蚌类物种数据进行冗余分析(Redundancy analysis, RDA)显示环境因子中水深(WD)、透明度(SD)、水温(WT)和总氮(TN)均显著影响排序结果(表 4)。
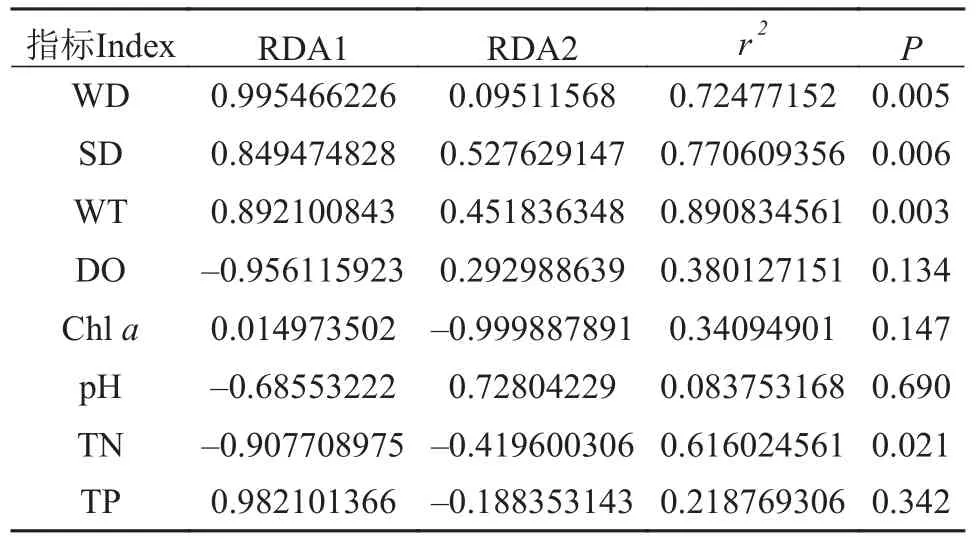
表4 环境因子对蚌类群落结构的差异性检验Tab. 4 Difference test of environmental factors on community structure of mussels
各蚌类物种与环境因子之间的相关性Heatmap分析表明: 河蛏蚌、扭状矛蚌、矛形楔蚌、圆头楔蚌、短褶矛蚌、橄榄蛏蚌、鱼尾楔蚌和背角无齿蚌均与溶解氧和总氮呈显著或极显著负相关,与水深呈显著或极显著正相关。大多数的蚌类物种与叶绿素a、透明度和水温之间呈显著或极显著正相关。蚌类与酸碱度和总磷的相关性不大(图 6)。
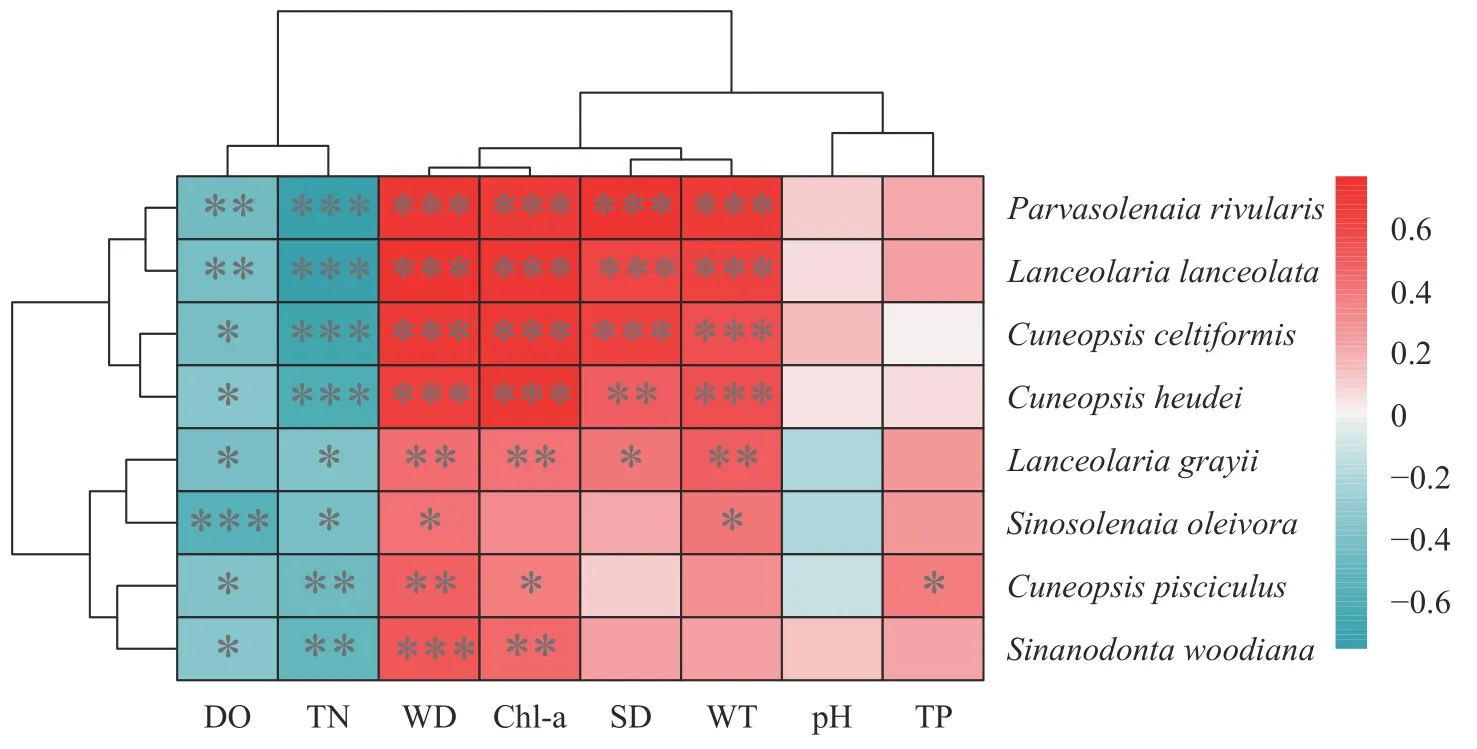
图6 蚌类物种与环境因子的相关性Heatmap分析Fig. 6 Heatmap analysis of correlation between mussel species and environmental factors
3.1 基于环境DNA高通量测序的鄱阳湖蚌类物种组成
本研究利用环境DNA宏条形码技术在鄱阳湖共注释到蚌类33种, 而传统方法共采集蚌类18种,且环境DNA注释到的蚌类物种覆盖了传统方法采集到的所有物种。这说明环境DNA宏条形码技术在蚌类的监测中分辨率高于传统方法的。这与陈金萍的调查结果一致[18]。Prié等[16]在其他双壳类的研究中也得出类似的结果。本研究注释到的33种蚌类中有30种是鄱阳湖历史记录种, 还有3种鄱阳湖非记录种: 扁棱蚌P. tenuis和鸭无齿蚌A. anatina和S. fukudai(该物种主要分布在韩国, 故没有找到相应的中文名字), 原因可能是本地数据库不完整造成的假阳性。如扁棱蚌、鸭无齿蚌和S. fukudai代表的OTU在本地数据库中注释到的分别是蚶形无齿蚌、扭状矛蚌和背角无齿蚌, 序列相似度分别为96.43%、95.53%和96.43%; 而在公共数据库中注释到的分别是扁棱蚌、鸭无齿蚌和S. fukudai, 序列相似度分别为97.32%、97.30%和97.32%。利用环境DNA宏条形码技术进行物种鉴定时, 所获结果的可靠性依赖于参考数据库的完整性和质量[26,27]。
本研究统计相对序列丰度高的物种分别为橄榄蛏蚌和圆顶珠蚌等, 而在传统调查中生物量高的物种分别为洞穴丽蚌和扭状矛蚌等(表 2)。虽然相对序列丰度受物种丰度的影响, 在一定程度上相对序列丰度体现了物种在环境中可能具有的丰度相互关系[28], 但二者的相关性不太确定[29,30], 而且环境中物种DNA的释放、运输和降解, 到环境样本的采集与提取、目的片段的扩增和高通量测序, 这些因素都会影响相对序列丰度与物种丰度的关系[31]。橄榄蛏蚌有历史生物量的记录, 且橄榄蛏蚌的生物量不高[32]。本研究传统采样方法中没有采集到橄榄蛏蚌活体(只采到空壳), 但是环境DNA相对序列丰度却最高, 可能跟以下两个因素有关。一是跟传统方法采样所用的工具有关, 传统方法采样通常用彼得森采泥器或自制蚌耙, 而橄榄蛏蚌的生活习性不同于其他蚌类, 使用彼得森采泥器或自制蚌耙比较难采到; 二是引物偏好性导致的环境DNA相对序列丰度高。
本研究中很多的蚌类物种只可分辨母系来源的序列, 是因为数据库中父系数据的缺失。蚌类在繁殖期雄性个体将精子排到水中, 而雌性个体的成熟卵细胞则聚集于鳃水管内, 接着精子通过水流而进入雌蚌鳃水管, 完成卵细胞的受精过程[33]。在能分辩父系和母系来源的序列物种中, 繁殖期正逢采样期的物种通常父系reads多于母系的, 如背角无齿蚌和圆顶珠蚌[34]; 繁殖期不处在采样期的物种通常母系reads更多, 如橄榄蛏蚌[35]。
3.2 基于环境DNA高通量测序的鄱阳湖蚌类多样性分析
基于环境DNA宏条形码的鄱阳湖蚌类Alpha多样性显示冬季和春季的蚌类多样性水平差异不显著, 传统方法的研究也表明蚌类多样性水平季节差异不显著[36]。春季注释到的蚌类物种数多于冬季,这和多数蚌类所处的繁殖期恰好是本研究的采样期有关; 传统方法的采集显示冬季采集到的物种数多于春季, 这与冬季较低的水位有关。基于Bray-Curtis距离矩阵的Beta多样性分析显示鄱阳湖冬季和春季的蚌类群落结构差异显著, 这可能与这两个季节分别注释到的物种数不同有关。
水体样本注释到的蚌类物种数多于沉积物样本注释到的物种数, 水体样本蚌类Alpha多样性水平显著高于沉积物样本的, Beta多样性分析也显示水体样本和沉积物样本存在显著性差异。水体中环境DNA来源广泛, 释放进入水体的DNA因沉降作用而在沉积物中积累, 而沉积物又在外力(如垂直流、底栖生物扰动和人为干扰等)作用下再悬浮[37],向周围释放环境DNA, 使得水体中环境DNA浓度升高, 从而使得水体样本检出的物种种类和多样性值均高于沉积物样本。表层水和底层水注释到的蚌类物种数一样多, 表层水和底层水Alpha和Beta多样性均无差异, 这说明蚌类的环境DNA在水体中无垂直分布差异。表层水和底层水注释到的蚌类物种数均分别高于沉积物的, 且表层水和底层水注释到的蚌类物种分别完全覆盖沉积物的(表 1)。这些结果表明在进行蚌类的环境DNA研究时采水样比采沉积物效果好, 且表层水和底层水无显著差异。
3.3 基于环境DNA高通量测序的鄱阳湖蚌类物种与环境因子的冗余分析
本研究通过关联鄱阳湖蚌类物种信息和环境因子表明水深(WD)显著影响排序结果, 这和陈金萍的结果一致[18]。这与蚌类喜栖息在浅水区有关。过深的水位通过影响光照强度, 降低水体透光性, 从而影响生物的光合作用, 使得浮游生物的生产量降低[38], 使得蚌类食物来源减少。且随着水深增加, 溶解氧(DO)含量降低[39], 过低的溶解氧会对水生生物产生不利影响[40]; 而在水位过浅时, 蚌类暴露和被捕食的风险也将增加[41]。透明度(SD)、水温(WT)和总氮(TN)也是造成冬季和春季蚌类物种群落结构变化的重要环境因子。透明度是判断水体藻类多寡的最直观的指标[42], 蚌类的食物组成与水体藻类组成有紧密关系[43], 故透明度也是影响蚌类群落结构变化的因子之一, 传统方法对蚌类研究也得出相同的结果[12]。适宜的水温, 不仅可以提高蚌类摄食强度和新陈代谢强度[36], 还有利于浮游植物的生长与繁殖[44]。蚌类对氨的敏感性很高, 尤其是在其幼虫和幼体阶段[45,46]。Galbraith等[47]的结果表明: 蚌类物种丰富度的长期下降与氮浓度的增加有关, 故总氮(TN)也是显著影响蚌类物种群落结构的重要因子之一。也有研究报道鱼尾楔蚌与水温成正相关, 与溶解氧呈负相关, 这是因为鱼尾楔蚌的呼吸和排泄与水温息息相关, 在一定水温范围内, 随着水温的升高, 鱼尾楔蚌的耗氧率和排氨率均增加[48]。水温作为控制溶解氧分布和变化最重要的环境因子[49], 水温的增加会使溶解氧浓度降低[50,51]。
环境DNA宏条形码技术能够有效地检测蚌类物种, 提供关于其物种组成和群落结构的数据, 能发现传统方法中经常被低估的稀有物种或很难找到的物种。使用环境DNA宏条形码技术监测蚌类资源时采水样比采沉积物效果好, 且表层水和底层水无显著差异。完善的参考数据库可增加检测的可靠性, 同时不过度依赖分类学专家。但参考数据库的构建需要分类学专家的协助以确保正确的物种鉴定。在环境DNA研究的发展领域中读数和生物量之间的关系, 以及这些关系如何受到环境变量、特定环境DNA来源等的影响还是不清楚。随着环境DNA宏条形码技术的不断发展, 我们预计它将会越来越多地被用于描述水生生物的多样性, 为水生生态系统的保护和管理提供依据。
致谢:
感谢郭婷、吴陈慧孜、贾晨等在水样采集工作中提供的帮助; 感谢黄晓晨老师和代雨婷在数据分析方面提供的帮助。
猜你喜欢鄱阳湖条形码沉积物鄱阳湖水系之潦河水利水电快报(2022年7期)2022-07-18晚更新世以来南黄海陆架沉积物源分析海洋通报(2022年2期)2022-06-30渤海油田某FPSO污水舱沉积物的分散处理海洋石油(2021年3期)2021-11-05创意条形码少年文艺·开心阅读作文(2021年8期)2021-09-05水体表层沉积物对磷的吸收及释放研究进展河北环境工程学院学报(2021年1期)2021-03-19《鄱阳湖生态系列插画》现代出版(2019年6期)2020-01-14从条形码到二维码小学科学(学生版)(2019年5期)2019-05-21从条形码到二维码少儿美术(快乐历史地理)(2019年11期)2019-04-20条形码大变身小学生导刊(2017年13期)2017-06-15鄱阳湖好风光老友(2017年4期)2017-02-09 相关热词搜索:环境,样本,监测,






