利用mRNA-seq和Ribo-seq技术研究低氧条件下SW620细胞的基因表达特征
时间:2023-02-27 10:30:08 来源:千叶帆 本文已影响人
梅 勇,梁绮华,邓载安,汪劲松
(1.湖北师范大学生命科学学院 食用野生植物保育与利用湖北省重点实验室,湖北 黄石 435002;
2.深圳技术大学健康与环境工程学院,深圳 坪山 518118)
核糖体印迹测序(ribosome profiling, Ribo-seq)是由Ingolia等发明的一种对基因翻译组学研究的手段[1],可以对正在翻译的RNA进行选择性测序,可用于揭示基因翻译特征。其原理是利用翻译抑制剂(放线菌酮)阻止核糖体翻译,然后消化核酸,捕获和富集核糖体内被保护的免受核酸酶切割的短基因片段,这些小片段又被称为核糖体足迹(ribosome footprints,RFs)[2],通过对RFs的高通量测序获得正在翻译的基因信息和定量信息。此外,根据数据分析可以获得核糖体在每个转录本上的分布和密度,推断出起始密码子位置(包括非ATG起始)、密码子使用偏爱、上游ORFs (uORFs:开放阅读框)和翻译暂停景观等信息。Ribo-seq还能观察到单个转录物的全局3-nt周期性,可发现小ORFs[3].核酸序列生物信息学分析能选择与核糖体足迹一致的读数,将读数的起点或终点重新校准到核糖体的肽基位点(P位点),并对转录物、密码子、多密码子基序的读数进行定量[4]。
越来越多的实验和研究表明,低氧环境是实体瘤中心常常出现的现象,特别是结直肠癌[5]。低氧诱导因子1α(HIF1-α)是参与低氧转录激活的主要转录因子,在低氧条件下,脯氨酰羟化酶和天冬酰胺酰羟化酶被抑制,从而抑制关键HIF1-α的羟基化,阻断泛素-蛋白酶体系统对HIF1-α的降解作用,使得HIF1-α的快速积累以适应低氧环境[6],HIF1-α通过激活下游靶基因来参与癌细胞的增殖、凋亡、血管生成和转移以及放化疗敏感性等生理病理过程[7]。有临床数据证明,肿瘤内低氧环境或基因突变引起的HIF1-α高表达与许多类型癌症患者的死亡率增加有关[8]。然而,低氧条件下的细胞整体基因表达和翻译特征仍不清楚[9]。
细胞中蛋白质水平取决于基因转录水平、mRNA翻译水平和蛋白质降解水平及其他因素[10]。为了研究低氧条件下结直肠癌细胞基因表达特性和调控机制,我们利用CoCl2化学模拟低氧条件来处理SW620细胞,使用mRNA-seq和Ribo-seq方法检测CoCl2处理前后转录和翻译水平的变化,并通过比较基因的翻译效率来寻找低氧条件下调控的基因。本项目的研究为低氧调控结直肠癌细胞的基因表达提供了一个新的角度。
1.1 试剂、细胞株和仪器
胎牛血清购自美国Gibco公司(10099141C);
DMEM-高糖培养基(SH30022.01)、PBS(SH30256.01)、青霉素/链霉素(SV30010)、胰酶(SH30042.01)均购自美国Hyclone公司;
HIF1-α抗体(3716S)购自美国CST公司;
GAPDH抗体(60004-1-Ig)、羊抗兔IgG(SA00001-2)、羊抗鼠IgG(SA00001-1)购自武汉三鹰生物技术有限公司;
GLUT3抗体(bs-1207R)、AARE抗体(bs-5983R)购买自北京博奥森生物技术有限公司;
RIPA细胞裂解液(R0278-500ML)、CoCl2(C8661-100G)购自Sigma公司;
RNA提取试剂盒(AG21017)、反转录试剂盒(AG11706)均购自湖南艾科瑞生物工程有限公司;
引物均购自华大基因;
人结肠直肠腺癌SW620细胞系购自南京科佰生物科技有限公司;
细胞培养箱购自美国Thermo 公司;
凝胶成像系统购自上海天能公司;
TAdvanced 96G PCR 扩增仪购自德国Biometra公司;
QuantStudio 6 Flex实时荧光定量PCR仪购自美国Thermo公司。
1.2 方法
1.2.1 细胞复苏及培养 SW620细胞快速从液氮罐取出,放入37℃恒温水浴锅中轻轻晃动融化,细胞冻存液转移到含5 mL(10%胎牛血清和1%双抗)DMEM-高糖培养基的25 mm2培养瓶中,在37℃含5% CO2细胞培养箱中培养。
1.2.2 CoCl2化学模拟低氧处理 配制200 mM CoCl2母液,SW620细胞生长状态良好的情况下,分别加入终浓度为0、50、100、200 μM的CoCl2溶液,细胞培养2~3天。
1.2.3 Western blot 加入适量RIPA溶液提取细胞的总蛋白,利用BCA方法测定蛋白浓度;
样品加入上样缓冲液(Loading Buffer(5×),在100 ℃金属浴煮样5 min;
每孔加入15 μg蛋白进行SDS-PAGE电泳;
再将蛋白转移至PVDF膜,在5 %脱脂奶粉溶液中封闭1 h;
一抗孵育(GAPDH 1∶20000;
HIF1-α 1∶1000;
SLC2A3 1∶1000;
APEH 1∶1000),4℃过夜;
二抗孵育(羊抗兔IgG 1∶3000;
羊抗鼠IgG 1∶5000),室温孵育45 min;
ECL化学发光显影并拍照。
1.2.4 mRNA-seq和Ribo-seq 实验的样品准备 mRNA-seq和Ribo-seq实验由广州表观生物科技有限公司完成。1)mRNA-seq文库构建:用Trizol试剂提取总RNA,mRNA-seq根据VAHTSTM Stranded mRNA-seq Library Prep Kit for Illumina v2 进行制备。2)Ribo-seq文库构建:0 μM和200 μMCoCl2处理的SW620细胞,含放线菌酮(Cycloheximide)PBS溶液洗涤,刮下细胞转移到离心管中离心5 min,去上清,重复一次。细胞裂解物先经过核酸消化,将ribosome bounding RNA与游离mRNA分离。然后富集核糖体-mRNA复合物,得到RPFs.去除rRNA并用PAGE胶纯化后,得到目的RNA片段。在两端加5’和3’接头进行末端修饰。反转录扩增得到的cDNA进行PAGE胶纯化。
1.2.5 测序方法及数据分析 1)将上述方法准备好的样品,利用Bioptic Qsep100 Analyzer对mRNA-seq和Ribo-seq文库大小分布进行质检。2)测序: 在Illumina NovaSeq 6000平台PE150进行测序。3)数据分析:首先对原始数据进行质量控制处理,获取Clean data,利用 Fast QC 分析质量分布,碱基含量分布,重复测序片段比例等测序的数据质量;
利用 hisat2 软件将过滤后的mRNA-seq和Ribo-Seq的reads数据与参考基因组比对;
使用htseq-count对mRNA-seq上的reads数进行统计,使用feature Counts软件计算ORF区内Ribo-seq水平的reads数,将其转换为FPKM (Fragments Per Kilobase Million)值;
使用 DEseq2 软件通过log2FC>0条件筛选差异翻译效率基因;
筛选出的差异翻译效率基因采用 Fisher 检验计算筛选出显著性GO和KEGG.
1.2.6 RNA提取和RT-qPCR 根据试剂盒说明书,提取总细胞RNA并逆转录成cDNA.实时荧光定量PCR仪反应程序为:95 ℃ 10 min,1个循环;
95 ℃ 60 s,60 ℃ 30 s,40个循环。GAPDH作为内参,用2-ΔΔCt法计算相对基因表达水平,每个样品三个重复孔(所有使用的引物如表1所示)。

表1 引物序列信息
1.2.7 统计学分析 通过Origin 9.0统计绘图软件进行数据处理,采用独立样本t检验比较两组间统计学差异,*p<0.05,**p<0.01,***p<0.001.
2.1 CoCl2化学处理SW620细胞模拟低氧条件
CoCl2化学处理是一种常用的化学模拟低氧处理试剂,CoCl2是羟化酶活性的竞争性抑制剂,可以抑制低氧因子HIF1蛋白的羟基化,减少HIF1与pVHL结合来达到稳定HIF1蛋白目的[11,12]。在本实验中,我们分别利用0 μM, 50 μM, 100 μM, 200 μM的CoCl2处理SW620细胞,处理后的细胞利用Western blot技术检测不同条件下HIF1-α的蛋白含量,实验结果显示SW620细胞随CoCl2浓度升高,HIF1-α蛋白的表达逐渐增加,在200 μM表达量最高(图1)。因此,我们选用了200 μMCoCl2处理的SW620细胞进行mRNA-seq和Ribo-seq实验。

图1 CoCl2处理SW620细胞HIF1-α蛋白含量
2.2 mRNA-seq和Ribo-seq整体数据特征分析
为了比较低氧条件对SW620细胞的转录水平和翻译水平的影响,我们将200 μM的CoCl2处理的SW620细胞分别分成两组,一组提取总RNA,用于mRNA-seq采集数据,一组用放线菌酮处理,按照Ribo-seq实验流程获得采集数据。数据显示,在mRNA-seq中,0 μM和200 μMCoCl2处理的样本的Total Read分别为86226028和93874676,Q30_rate分别为0.978和0.978;
Ribo-seq实验中,Total Read分别为95491377和92705635,Q30_rate分别为0.954和0.967(表2)。这些数据显示mRNA-seq和Ribo-seq采集的数据总量和质量可满足下游分析。
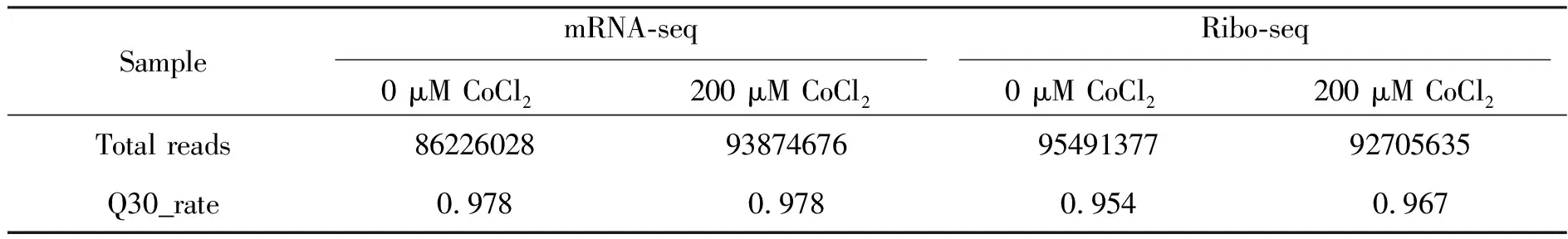
表2 mRNA-seq和Ribo-seq的样本序列质控统计结果
在Ribo-seq数据中,我们发现0 μM和200 μMCoCl2处理的样本RFs主要分布在25~34 nt(图2a, 2c)。利用riboWaltzR对25~34 nt的评估RFs在RNA上的3碱基的周期性分布,如图2b, 2d看出,横坐标为距离start-codon or stop-codon 的距离,纵坐标为比对上的reads.0 μM和200 μMCoCl2处理的SW620两组的25~34 nt的RFs符合3 nt特征,这也说明本研究中Ribo-seq数据质量可靠。

图2 SW620 在0 μM和200 μM CoCl2处理下核糖体的RFs长度分布(a,c)以及25~34 nt长度的RFs在翻译起始位点后50 nt和终止密码子前50 nt的3 nt特征(b,d)
2.3 CoCl2化学模拟低氧处理对基因转录水平及翻译水平的影响
为了分析每个样本中的基因转录水平与翻译水平之间的关系,我们对每个样本的mRNAs和核糖体结合mRNAs的丰度采用TPM (Transcripts Per Kilobase Million)方法来进行均一化处理,然后计算每个基因的log10(TPM+1)值,通过相关性分析发现,两个样本中基因的转录水平和翻译水平的皮尔森相关系数(Pearson correlation)分别为0.77和0.76(图3a,3b),说明在我们这两个样本基因的表达水平和翻译水平具有强的正相关性。

图3 0 μM和200 μM CoCl2处理下转录和翻译水平上log10(FTM+1)关系(a,b)以及200 μM CoCl2处理后转录或翻译水平的差异表达基因数量(c)和转录和翻译水平上的基因关系(d)
根据mRNA-seq数据显示在0 μM CoCl2处理检测到24 613个基因,200 μM CoCl2处理检测到24 321个基因。与0 μM CoCl2处理相比,200 μM CoCl2处理后的细胞有118个基因上调,119个基因下调。根据Ribo-seq数据我们发现0 μM CoCl2处理下检测到6 829个基因,200 μM CoCl2处理后检测到4 941个基因。与0 μM CoCl2处理相比,200 μM CoCl2处理后的细胞中有254个基因翻译水平上调,646个基因翻译水平下调(图3c)。其中,4个基因在转录水平和翻译水平都上调,16个基因在转录水平和翻译水平都下调(图3d)。上调的基因分别为NDRG1、CXCL8、MIR22HG、SLC2A3,下调的基因分别为PTK7FIBP、PRSS2、CHCHD10、APEH、BEX3、RAB13、DNPH1、ATF6B、FAM210B、TSTD1、STRA6、SCRN2、IGFBP6、VWA1、TMEM176A.KEGG分析结果显示,低氧条件下,翻译效率差异基因主要富集在10个信号通路:癌症中蛋白聚糖,中性粒细胞胞外陷阱形成,酗酒,病毒致癌作用,系统性红斑狼疮,细胞衰老,补体系统,FOXO信号通路,ECM受体相互作用,膀胱癌(图4d)。

图4 差异翻译效率基因GO细胞组分(a)、分子功能(b)和生物过程(c)分析结果以及差异翻译效率基因KEGG排名前10的富集结果(d)
2.4 RT-qPCR和WB验证基因和蛋白表达
为了探究预测的mRNA-seq和Ribo-seq数据是否与蛋白质水平相匹配。我们从转录水平和翻译水平变化一致的基因中,挑选了部分基因进行了RT-qPCR(NDRG1、SLC2A3、APEH、TMEM176A、FAM210B)和Western blot(SLC2A3、APEH)实验。RT-qPCR结果显示,在低氧条件下,NDRG1和SLC2A3在转录水平上调(图5a, b),APEH、TMEM176A、FAM210B在低氧条件下转录水平下调,但SLC2A3不显著。Western blot结果显示SLC2A3蛋白质表达水平没有显著变化,APEH蛋白质水平降低(图5c)。这些结果显示暗示有的基因的蛋白水平可能与基因的转录水平和翻译水平不一致。

图5 0 μM和200 μM CoCl2处理下HIF1-a、NDRG1、SLC2A3基因表达(a),APEH、TMEM176A、FAM210B基因表达(b)和APEH、SLC2A3蛋白表达(c)情况
低氧是肿瘤组织的重要特征之一,但是低氧如何调控基因表达的研究仍需要深入研究。本项目中,我们利用mRNA-seq和Ribo-seq技术分析了CoCl2化学模拟低氧处理SW620细胞后基因在转录水平和翻译水平变化,并对基因的翻译效率进行了分析。据我们所知,这项研究是率先利用mRNA-seq和Ribo-seq技术研究低氧条件下SW620基因表达特征的报告,这些数据揭示了低氧条件下SW620的基因表达模式,将有助于进一步认识结直肠癌细胞的特征。
多种方法可以实现细胞的体外低氧培养,包括低氧培养箱,低氧培养装置,化学处理等。CoCl2还可以模拟建立体外Caco-2结直肠癌细胞和MCF-7乳腺癌细胞的缺氧模型[13]。在本研究中,对HIF1蛋白的Western blot分析发现CoCl2处理SW620细胞也可使HIF1-a蛋白含量升高,也证明了CoCl2处理SW620细胞模拟低氧环境是可行的。
本项目中,我们分别采集了CoCl2处理的SW620细胞的转录组学(mRNA-seq)和翻译组学(Ribo-seq)数据。每个处理组的转录组学totalread超过100M,QC30超过了95%,可以判断两组转录组学数据量和质量可满足下游分析的。在翻译组学数据中,RFs长度主要在25~34 nt之间,符合预期的核糖体保护的RNA片段(RFs)的长度在28 nt左右的特征。翻译过程中,核糖体在可翻译的RNA以密码子长度(3 nt)为单位移动,完成一次肽段延伸[14]。因为核糖体在密码子第一碱基位置停留时间最长,因此RFs 比对位置对应密码子第一个碱基的比例通常最高,而第二第三个碱基都偏低,RFs片段应该在翻译的RNA的开放阅读框(ORF)中呈现数据呈现“高-低-低”三碱基周期模式,即3 nt特征。我们的Ribo-seq实验中的25~34 nt 的RFs也符合这些特征。由此,我们认为本项目中的转录组学(mRNA-seq)和翻译组学(Ribo-seq)数据质量好。
组学技术发展有助于我们对基因表达在不同水平的进行研究,为我们了解基因表达调控提供思路。本项目中,我们利用两组转录组学和翻译组学中的RNA丰度进行了相关性分析,研究发现两组中的基因在转录水平和翻译水平都呈强相关性。基因差异表达分析发现翻译水平上的变化基因数目大于转录水平上变化的基因。同时,我们发现翻译水平被抑制的基因多于增强的基因。这暗示着低氧条件可抑制基因的表达,这与Lei等人在玉米中的研究发现低氧抑制基因翻译的现象相符合[15]。
我们对翻译水平和转录水平上变化一致的基因进行了统计,这暗示着低氧条件可以对这些基因的转录水平和翻译水平都存在着调控。NDRG1 (N-myc下游调节基因1)是一种与肿瘤侵袭和迁移密切相关的多功能基因,其通过G1/S期阻滞细胞周期来抑制Caco2细胞的增殖,且NDRG1过度表达时,侵袭和迁移的强度降低[16]。多个研究组表明该蛋白受HIF1-α的上调[17,18]。SLC2A3是编码葡萄糖转运蛋白的溶质载体家族,也称为GLUT3,介导葡萄糖从细胞外转运到细胞内,在癌细胞的代谢重编程中起关键作用[19]。SLC2A3基因的上调与结直肠癌患者的OS和DFS降低有关[20]。Taniguchi-Ponciano等在新冠肺炎重症患者中发现HIF1a及其下游与碳水化合物代谢相关的SLC2A3基因表达都增加[21]。但是在本研究中,我们发现SLC2A3的转录水平受到低氧诱导表达但不显著,蛋白水平也没有显著变化。这可能是由于不同的细胞组织低氧对SLC2A3诱导表达的程度不同导致的,也有可能是由于不同细胞对SLC2A3蛋白稳定性调控不同。APEH(N-酰肽水解酶)是一种细胞溶质酶,又称氨基酸释放酶(AARE),属于丝氨酸肽酶的脯氨酰寡肽酶家族,在泛素系统以及蛋白质分解过程中起重要作用[22]。Palmieri等发现,与健康对照相比,在阿茨海默症(AD)样品中APEH活性显著降低[23]。TMEM176A(人跨膜蛋白176A)的表达诱导H1299和H23细胞G2/M期阻滞导致细胞凋亡,并抑制集落形成、细胞增殖、迁移和侵袭,其通过抑制ERK1/2信号来抑制肺癌生长[24]。FAM210B一种线粒体外膜蛋白的癌症进展抑制基因,FAM210B的低表达导致线粒体呼吸能力增加和糖酵解减少,从而激活EMT并增强迁移和侵袭特性,FAM210B缺失与体内和体外存活率降低和转移增强显著相关[25]。
翻译效率可以反映基因在翻译水平上的精细调节。为了研究低氧对基因表达调控机制,我们分析了低氧条件下的基因翻译效率的差异基因。KEGG分析发现低氧条件下最主要影响就是癌症中蛋白聚糖,性粒细胞胞外陷阱形成,酗酒,病毒致癌作用,系统性红斑狼疮,细胞衰老,补体系统,FOXO信号通路,ECM-受体相互作用,膀胱癌信号通路中的基因翻译效率。其中蛋白聚糖信号通路是富集程度最高的一个通路,该通路中的26个基因的翻译效率受低氧调控,之前的研究表明相关的基因有助于肿瘤生长基质的形成,影响细胞—细胞和细胞—基质相互作用以及肿瘤细胞的信号转导,还调节肿瘤细胞的表型和肿瘤基质血管生成[26,27]。Zhang等人在卵巢癌细胞中发现低氧可调节蛋白聚糖信号通路[28]。本研究揭示了低氧调控相关的基因的表达可能是涉及翻译水平的调控。
本文联合了转录组学和翻译组学对低氧条件下结直肠癌细胞SW620的转录水平进行了分析,发现了一系列在转录水平和翻译水平变化一致的基因,更重要的是通过比较不同条件下基因翻译效率发现,低氧可调控多个信号通路的基因翻译效率,为阐述低氧调控基因表达机制提供新的角度。
猜你喜欢 核糖体低氧组学 低氧阈刺激促进神经干细胞增殖分化山东第一医科大学(山东省医学科学院)学报(2022年8期)2022-12-07核糖体成熟因子RimP、Era和RimJ的研究进展内蒙古民族大学学报(自然科学版)(2022年2期)2022-11-22影像组学在肾上腺肿瘤中的研究进展昆明医科大学学报(2022年3期)2022-04-19近三百年来长江口泥质区沉积环境变化及与低氧关系的初步分析海洋通报(2021年1期)2021-07-23东莨菪碱中毒大鼠的代谢组学昆明医科大学学报(2021年4期)2021-07-23间歇性低氧干预对脑缺血大鼠神经功能恢复的影响天津医科大学学报(2021年3期)2021-07-21影像组学在核医学影像中的应用进展智慧健康(2021年33期)2021-03-16蛋白质组学技术在胃癌研究中的应用天津医科大学学报(2021年1期)2021-01-26核糖体生物合成与肿瘤的研究进展肿瘤防治研究(2020年5期)2020-07-09例析翻译过程中核糖体移动方向的判断生物学教学(2019年9期)2019-09-23 相关热词搜索:低氧,条件下,技术研究,






